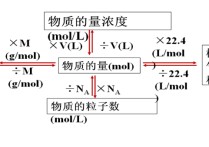
生物信息学导师有哪些 华科的田岩导师
武汉大学生命科学院遗传学导师信息,南开大学生科院生物工程专业硕士有哪几个导师,钟扬的复旦大学教授,求教生物信息学研究生学长,怎么选择导师?华科大薛宇导师,生物信息学领域中,有哪些不得不知道的大牛教授/研究者。
本文导航
安徽农业大学生命科学院教师介绍
模式植物遗传学副教授:章志宏
章志宏,男,武汉大学生命科学学院教授、博士生导师。专业与研究方向为植物基因组学与分子改良。
1979-1983年,华中农业大学农学系本科生,毕业后获理学学士学位
1983-1988年,湖北省农业厅粮食生产处工作,从事水稻生产技术示范推广
1988-1991年,武汉大学遗传学硕士研究生,毕业后获硕士学位、并留校任教
1992年晋升讲师,2002年晋升副教授,2004年获遗传学博士学位
2008年晋升教授
利用植物基因组学的原理、方法和数据库,解析水稻重要性状(产量、品质和抗性等)的遗传基础,揭示重要性状的遗传网络;研究基因互作、基因与环境互作在性状发育中的作用;发掘优异功能新基因,通过分子标记辅助选择和基因工程等手段,进行水稻品种的分子改良,培育水稻新品种。
群体与数量遗传学教授 :胡中立
职称:教授、博士生导师
学位:博士
博士学位授予学校:武汉大学
专业:遗传学
研究方向
1、植物资源生物学与生物技术:植物资源遗传与改良,植物资源开发利用。
2、植物分子细胞遗传学:植物分子核型分析,染色体分拣及专性文库的构建与利用。
3、生物信息学和数量遗传:统计基因组学与QTL分析,植物基因与基因组进化分析,基因组作图。
科研项目
2000年以来曾先后主持及完成国家级科研项目6项,省、部级科研项目10项。
南开大学微生物研究生排名
现有一级学科:生物学(0710)
下属二级学科包括以下8个:
植物学(071001)
动物学(071002)
微生物学(071005)
遗传学(071007) 细胞生物学(071009)
生物化学与分子生物学(071010) 生态学(071012)
生物信息学(071020)另有高分子化学与物理(070305)(教育部生物活性材料重点实验室)是属于化学一级学科下的二级学科
抱歉。没有找到生物工程专业
下面是我找到的老师
饶子和
性 别:男
职 称:教授
所属部门:生物化学和分子生物学系
招生专业:
研究方向:
::教师简介::
分子生物物理和结构生物学家,中国科学院院士,第三世界科学院院士,全国人大代表,中国科协常委,中国生物物理学会理事长,国际纯粹与应用生物物理联合会(IUPAB )理事会执行理事。
主要从事与重要病毒和肿瘤相关的蛋白质结构、功能以及创新药物的研究,取得了一系列重要的原创性的成果,在SARS病毒、禽流感病毒和膜蛋白的三维结构与功能的研究中做出了突出贡献。在国际学术刊物上发表研究论文218篇,其中Nature/Cell系列论文13篇,包括Nature 3篇,Cell 2篇,被引用超过2000次,同时申请并获发明专利6项。
王磊
性 别:男
职 称:教授
所属部门:微生物学系
招生专业:微生物学、生物化学、生物信息学
研究方向:微生物遗传与进化、进化基因组学、微生物表面抗原功能基因
董金堂
性 别:男
职 称:教授
所属部门:遗传学和细胞生物学系
招生专业:遗传学、分子生物学、细胞生物学
研究方向:肿瘤发生和发展的分子机制研究,涉及发现和鉴定肿瘤相关基因,并对个别基因进行生化和分子通路的研究。
钟扬校长
湖南新宁人,1964年5月出生。中国科学技术大学少年班毕业,无线电电子学工学学士;日本国立综合研究大学院大学(The GraduateUniversity for Advanced Studies)生物系统科学博士。1984 - 1999年任中国科学院武汉植物研究所研究实习员、助理研究员、副研究员(1992)、研究员(1996)、副所长(1997);1992-1998年曾在University of California-Berkeley和Michigan State University进行合作研究4年;2002-2006年曾两次任日本文部科学省统计数理研究所外国人客员教授;2000年起任复旦大学生命科学学院教授,植物学和生物信息学博士生导师。现为复旦大学生命科学学院常务副院长,生物多样性与生态工程教育部重点实验室副主任,上海生物信息技术研究中心副主任;兼任北京大学理论生物学中心教授、西藏大学教授等。中国生物物理学会生物信息学与理论生物物理学专业委员会主任,中国植物学会系统与进化植物学专业委员会副主任。 2001年获中国高校自然科学一等奖 (排名第二)2015年9月17日,由中央电视台和光明日报社联合主办的2015“寻找最美教师”大型公益活动“被推选为活动“特别关注教师”。 [1] Jung, S., Perkins, S., Zhong, Y., Pramanik, S., Beaman, J. H. 1995. A new data model for biological classification. Computer Applications in Biosciences (CABIOS) 11: 237-246.[2] Zhong, Y., Jung, S, Pramanik, S, Beaman, J H. 1996. Data model and comparison and query methods for interacting classifications in a taxonomic database. Taxon 45(2): 223-241.[3] Zhong, Y., Meacham, C. A., Pramanik, S. 1997. A general method for tree-comparison based on subtree similarity and its use in a taxonomic database. BioSystems 42: 1-8.[4] Zhong, Y., Luo, Y., Pramanik, S., Beaman, J. H. 1999. HICLAS: a taxonomic database system for displaying and comparing biological classification and phylogenetic trees. Bioinformatics 15(2): 149-156.[5] Zhong, Y., Shi, S. H., Tang, X. H., Huang, Y. L., Tan, F. X., Zhang, X. Y. 2000. Testing relative evolutionary rates and estimating divergence times among six genera of Rhizophoraceae using cpDNA and nrDNA sequences. Chinese Science Bulletin 45: 1011-1015.[6] Shi, S., Jin, H., Zhong, Y., He, X., Huang, Y., Tan, F., Boufford, D. E. 2000. Phylogenetic relationships of the Magnoliaceae inferred from cpDNA matK sequences. Theoretical and Applied Genetics 101: 925-930.[7] Sang, T., Zhong, Y. 2000. Testing hybridization hypotheses based on incongruent gene trees. Systematic Biology 49: 422-434.[8] Cui, X. H., Zhong, Y., Chen, J. K. 2000. Influence of a catastrophic flood on densities and biomasses of three plant species in Poyang Lake, China. Journal of Freshwater Ecology 15: 537-541.[9] He, Z. C., Zhang, X. Y., Zhong, Y., Ye, L. 2000. Phylogenetic relationships of Actinidia and related genera based on micromorphological characters of foliar trichomes. Genetic Resources and Crop Evolution 47: 627-639.[10] Wang, J. B., Wang, C., Shi, S. H., Zhong, Y. 2000. ITS regions in diploids of Aegilops (Poaceae) and their phylogenetic implications. Hereditas 132: 209-213.[11] Wang, J. B., Wang, C., Shi, S. H., Zhong, Y. 2000. Evolution of parental ITS regions of nuclear rDNA in allopolyploid Aegilops (Poaceae) species. Hereditas 133: 1-7.[12] Zhong, Y., Zhang, L., Su, D. M. 2000. Collaborations tailored for bioinformatics projects. Science 290: 2074. (Letter)[13] Shi, S., Huang, Y., Zhong, Y., Du, Y., Zhang, Q., Chang, H., Boufford, D.E. 2001. Phylogeny of the Altingiaceae based on cpDNA matK, PY-IGS and nrDNA ITS sequences. Plant Systematics and Evolution 230: 13-24.[14] Wu, J., Liang, Y., Zhong, Y., Fu, C., Chen, J. 2002. Advances in phylogenetic studies of Nematoda. Chinese Science Bulletin 47: 10-15.[15] Huang, Y., Shi, S., Zhong, Y., Tan, F. 2002. A new method for preparation of template DNA for PCR from special plant materials. Chinese Science Bulletin 47: 725-727.[16] Shi, S., Zhong, Y., Huang, Y., Du, Y., Qiu, X., Chang, H. 2002. Phylogenetic relationships of the Rhizophoraceae in China based on sequences of the chloroplast gene matK and the internal transcribed spacer regions of nuclear ribosomal DNA and combined data set. Biochemical Systematics and Ecology 30: 309-319. 等。 【1】 钟扬, 陈家宽, 黄德世, 1990.。数量分类的方法与程序。武汉大学出版社。【2】 钟扬, 李伟, 黄德世, 1994。分支分类的理论与方法。科学出版社。【3】 钟扬, 张亮, 赵琼等, 2001。简明生物信息学。高等教育出版社。【4】 钟扬, 张文娟, 王莉,赵佳媛, 2005。基因计算。上海科技教育出版社。【5】 李伟, 钟扬编译, 1992。水生植被研究的理论与方法。华中师范大学出版社。【6】 M. Nei, S. Kumar著, 吕宝忠, 钟扬, 高莉萍等译, 2002。分子进化与系统发育。高等教育出版社。【7】 D. W. Mount著, 钟扬, 王莉, 张亮等译, 2003。生物信息学。高等教育出版社。【8】 J. D. Watson著, 钟扬, 沈玮, 赵琼, 王旭译, 2003。基因, 女郎, 伽莫夫。上海科技教育出版社。【9】 P. Reilly著, 钟扬, 李作峰, 赵佳媛, 赵晓敏译, 2005。林肯的DNA。上海科技教育出版社。
生物信息学研究生报考条件
学校现在研究生分导师一般是三种情况,一种是先录取后分导师,一种是录取前就要求先填报志愿。你的情况应该是属于前一种的,因为显然你现在导师还没有定。
一般如果导师已经定了的话,那就需要在暑假的时候先跟导师联系一下,因为有的导师比较喜欢在暑假的时候先让新生在自己实验室干干活,适应一下环境。象你这种导师最后还没有分配好的,最好就先别找了,除非你很确定人家最后会要你,否则很容易陷进学院人事斗争的陷阱里,作用反而适得其反。
选博导还是硕导的问题其实是个伪命题,因为这两者根本不在一个水平线上。博导会有比硕导多的多的研究课题和研究经费,在博导的手下不但活会比较多,而且外快也会比较多。
但如果博导有行政职务的话,除非你本来就是为了毕业找好工作,不在乎研究生期间能学到什么的话,不建议选。因为有行政职务的一般会比较忙,应酬比较多,你基本上学术上得不到多少指点,只能靠自己摸索了。
我当年就是图名气选了个博导,结果虽然活确实比较多,但基本上是干苦力的,感觉跟老板雇的员工一样,没学到什么.
华科的田岩导师
薛宇,男,1980 年6 月出生,分子细胞生物学博士,华中科技大学生命科学与技术学院教授、博士生导师,入选教育部“新世纪优秀人才支持计划”。主要研究方向为蛋白质共价修饰的生物信息学分析,近年来紧密围绕共价修饰底物和位点预测、修饰调控分析以及修饰组学数据处理等重要科学问题,采用由点及面、层层推进的研究策略,取得了阶段性的研究成果。与合作者设计了新颖的GPS (Group-based Prediction System, 分组预测系统) 系列算法,构建了多种预测共价修饰底物和位点的计算工具、软件和数据库;采用比较基因组学和统计学等方法,从蛋白质组层面系统分析了共价修饰潜在调控机制和功能,初步提出了本领域的标准化设计和分析流程;结合磷酸化组学数据,初步揭示遗传多样性改变蛋白质共价修饰状态,是其影响关键生物学过程和通路潜在分子机制;利用乙酰化组学数据,初步模拟人类乙酰化调控网络,提出由乙酰转移酶、底物和去乙酰酶组成的“三者关系”(Triplet relationship) 是构成人类乙酰化调控网络的基本单元。以主要作者 (第一或通讯) 身份发表SCI 收录论文近40篇,被他人引用~700次。应邀为InTech 出版社“Bioinformatics - Experimental Biology Systems”一书撰写一个章节。获国家自然科学基金、科技部和中国科学院等资助,获计算软件著作权登记证书四项。
生物信息学未来的看法
作者:知乎用户
链接:https://www.zhihu.com/question/42951437/answer/95466332
来源:知乎
著作权归作者所有。商业转载请联系作者获得授权,非商业转载请注明出处。
genomics或者说测序依然是很大的领域,其中的方向细分依然是很多的。genomics整体可以分成两大类,一类主要做方法,一类主要做生物。前者一般使用public data做一些方法,后者主要使用现有的技术研究生物问题。如果说大牛一般都在后一类里面。但是也会有很多不同的方向,比如研究rna的,dna的,基因调控的。所以最好根据自己关注的方向去找。
一个很直接找genomic方向大牛的方法就是看encode,roadmap和4D nuckesomes这些大genomic cobsortium的主要pi。当然也有很多牛人不在这些项目里面。下面简单说几个比较熟悉的,欢迎纠正和补充。
Eric Lander
Broad的创始人,人类基因组计划发起人之一,不过现在自己lab似乎不做大多了,但很多broad的paper还会挂名。
John Rinn
Broad的PI,研究lncRNA。最早发现了H19。
Michell Guttman
Eric Lander的学生,和John Rinn一起发现了lncRNA,在clatech独立不久。
Howard Chang
主要研究RNA,John Rinn的postdoc老板。
Michael Synder
研究基因调控,具体方向很多,组特别大。
Job Dekker
3D genome领域的开拓者,3C,5C和HiC的发明者。
Bing Ren
最早开发了chip-chip技术,研究enhancer,现在做更多3D genome。
Joe Ecker
最早是植物领域的大牛,后来转到做甲基化。
Chuan He
化学出身,之前做DNA甲基化,现在开创了RNA甲基化的新领域。
Mark Gersrein
比较少的主要做计算的大牛,各种network和system biology
Menolis Kellis
Eric Lander的学生,主要做计算也做实验,各种consortium都很活跃。
John Stam
名字很长很难写,主要做DHS,当然还有很多其他方向。
Shirely Liu
主要做计算,lab开发了MACS